Introducción
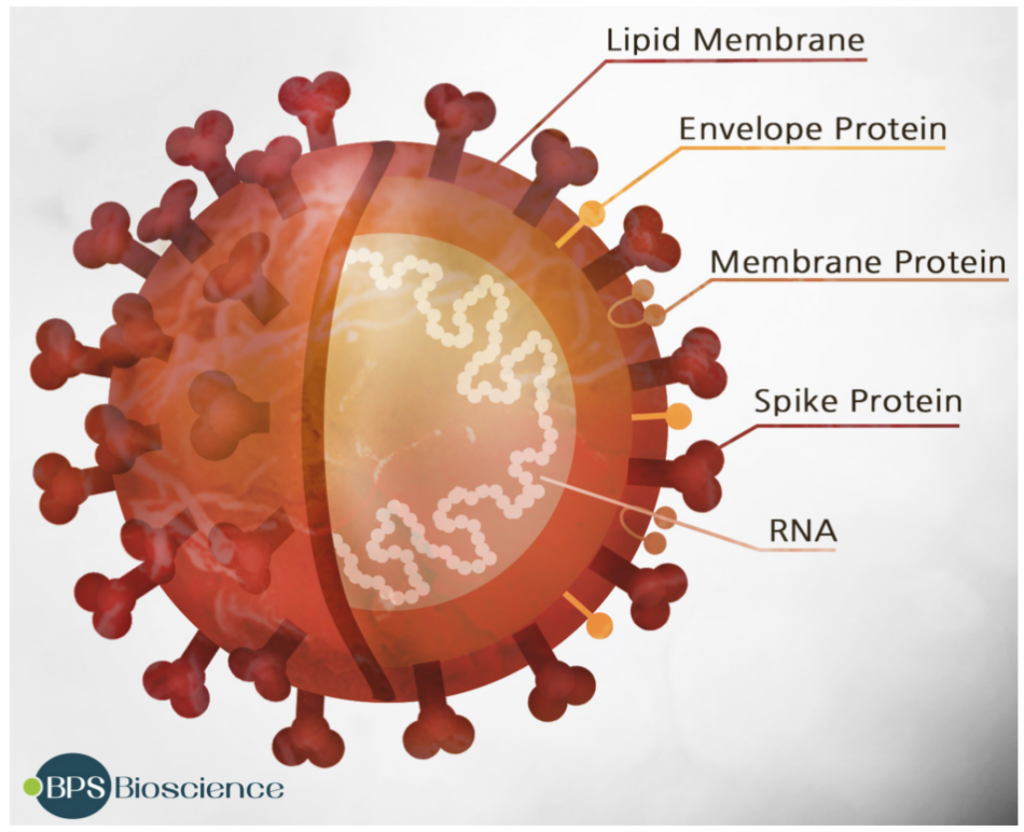
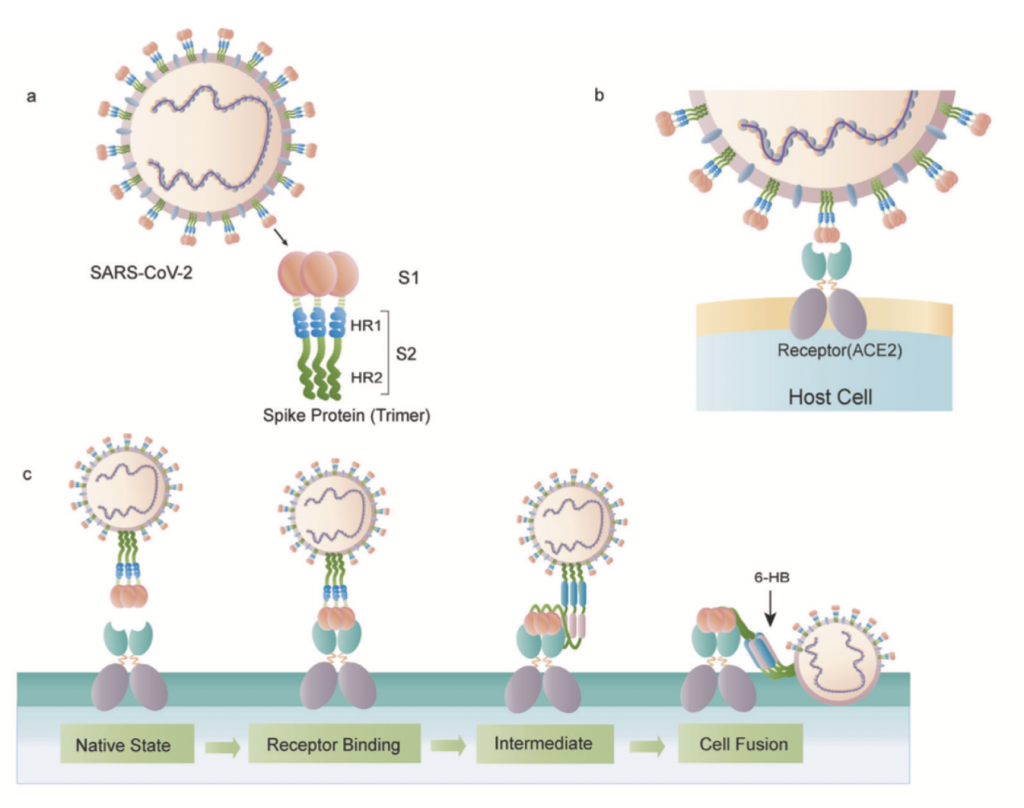
Desde la aparición del SARS-CoV-2 y su rápida propagación por todo el mundo, han surgido nuevas variantes con interés médico, inmunológico y epidemiológico pues estas podrían ser más patogénicas, evadir con más éxito la respuesta inmunitaria o hacer que las vacunas fueran menos efectivas. Habida cuenta que la proteína Spike (S) del coronavirus es crucial para la infección efectiva de una célula y, por consiguiente, la que media muchos de estos efectos negativos en la salud, todos los esfuerzos tanto en la profilaxis (vacuna) como en el diagnóstico y búsqueda de nuevas variantes preocupantes se centran en S. De hecho, estas nuevas variantes las empezamos a detectar pues algunas de las PCR fallaban en dar positivos en algunos de los fragmentos del gen S que se buscaban y porque las técnicas de secuenciación masiva están al alcance de muchos laboratorios de diagnóstico e investigación.
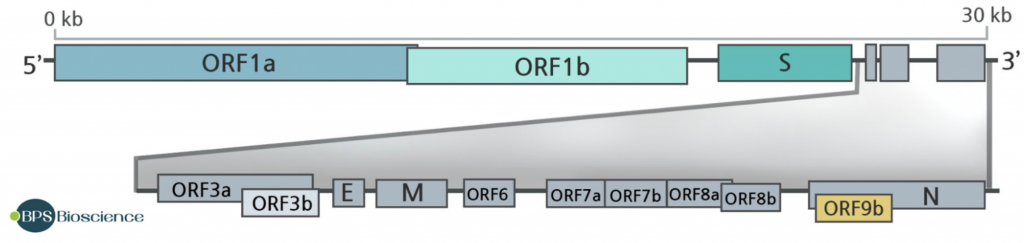
Pero hemos de tener presente, que el genoma del virus tiene 30.000 nucleótidos que codifican para 10.000 aminoácidos y que todos estos pueden mutar, no solamente los 1.273 aminoácidos de la proteína S. Además de las 4 proteínas estructurales, S, E (Envelope), N (Nucleocapside) y M (Membrane), el virus pose otras 16 proteínas no estructurales y 9 proteínas accesorias. Una vez dentro de la células, estas otras proteínas virales empiezan a jugar su papel vital para evitar la defensa intrínseca antiviral y que no se desencadene la respuesta inmune innata o para obligar a la maquinaria biosintética celular a enfocarse en la multiplicación y ensamblaje del virus. Y como hemos dicho esas proteínas también pueden mutar para ganar función y proporcionarle ventajas al virus, ya no solo en penetrar, sino en ser más infectivo.
Entre todo este conjunto de nuevas variantes posibles en este inmunoensayo nos vamos a centrar en las que juegan un papel en la batalla evolutiva para poder generar las variantes que escapen de los mecanismos dispuestos por la inmunidad innata. En particular podremos el foco a las mutaciones que intentan bloquear los sensores intracelulares que advierten a la célula que un virus ha logrado penetrar al citoplasma, los sensores que perciben ácidos nucleicos virales (RNA o DNA) intracelularmente. Este rol lo realizan muy rápidamente, en cuestión de minutos, provocando la producción y liberación de interferones de tipo I que de forma autocrina y paracrina alertan a todo el microambiente de la presencia de virus en ese tejido. Esta alerta hace que la célula entre en estado de actividad intrínseca antiviral y sintetice nuevas medidas para intentar degradar la información genética de virus y que esta se exprese o se puedan construir nuevos viriones. Pero el virus a lo largo de su evolución ha conseguido incluir en su genoma la codificación para proteínas que van a contrarrestar muy efectivamente esa defensa intrínseca antiviral. Todos los virus que parasitan a humanos tienen que intentar bloquear con contramedidas de más o menos intensidad esta respuesta antiviral. Y todos los humanos hemos de tener suficientes medidas antivirales para al final salir ganando la batalla. Es un fino equilibrio entre anfitrión y huésped tras muchos años de evolución.
Las características clínica de la covid-19 y la respuesta inmune al SARS-CoV-2 son bastante diferentes del resto de virus respiratorios. Ello podría justificar que ~20% de los infectados tengan distrés respiratorio agudo. Una de estas es la habilidad del virus para bloquear la respuesta inmunitaria innata, que sería la primera linea de defensa para eliminar eficientemente la carga viral del infectado. Aunque se ha descrito una híper-producción de citocinas de tipo pro-inflamatorio, el nivel de interferones de tipo I y III (IFN-β and IFN-ɣ), en plasma es más bajo de lo normal. Esta defensa basada en interferones es básica y de hecho los estudios genéticos a la búsqueda de susceptibilidades en la población a padecer la enfermedad más severa ha revelado que defectos en estos se asocian a una mayor morbimortalidad.
La participación de la mitocondria en la defensa intrínseca antiviral
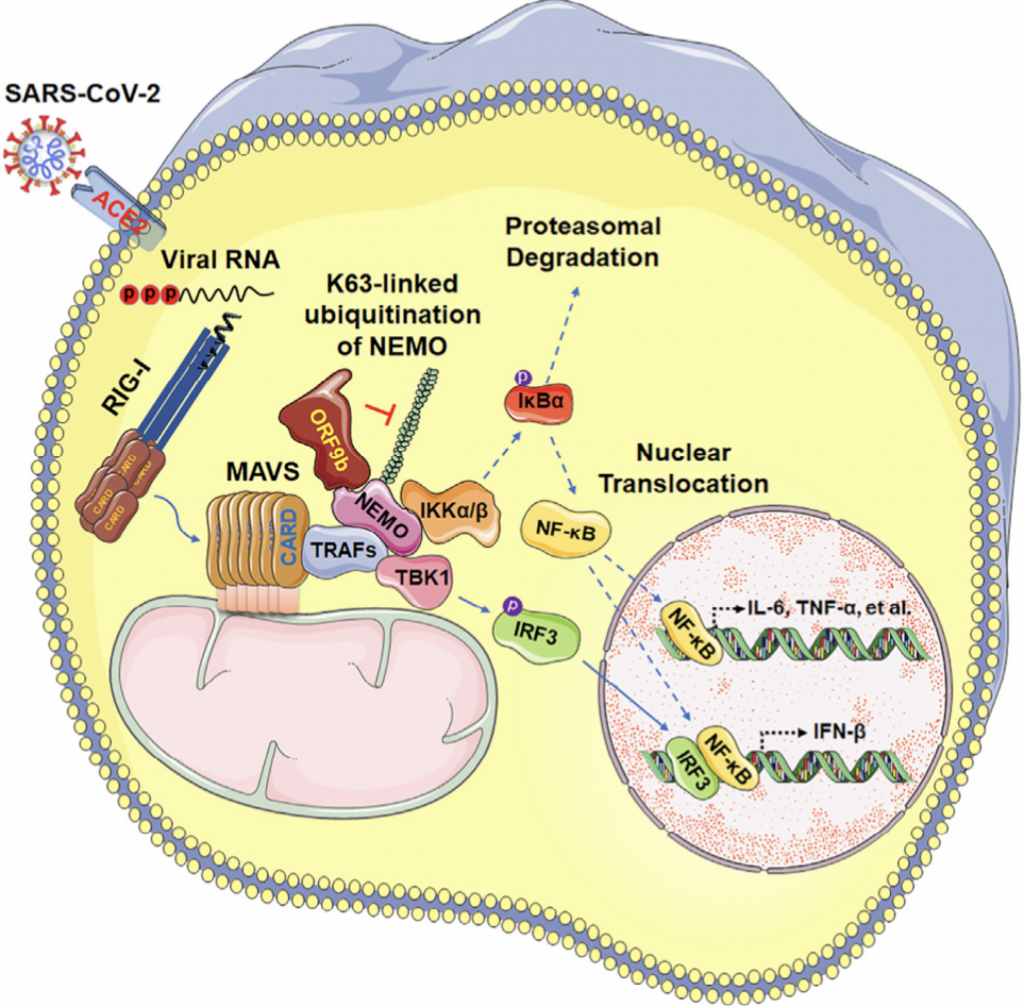
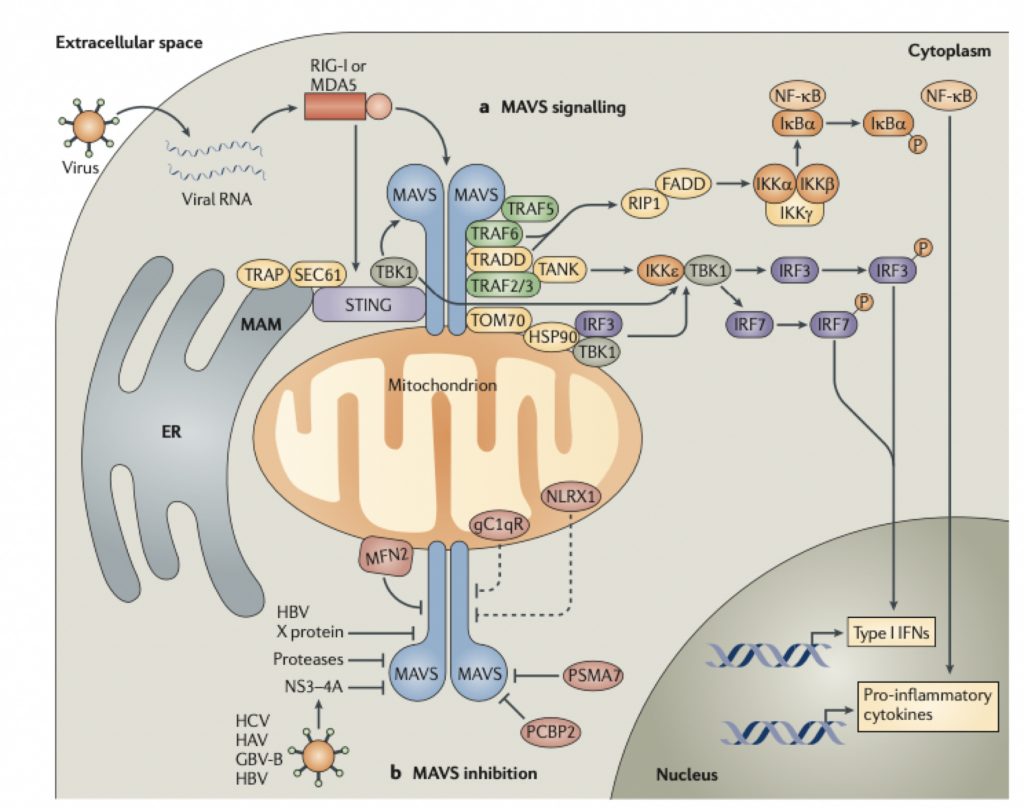
Tras la entrada en la célula al interaccionar la proteína S con el ectoenzima convertidor de angiotensina (ACE2) gracias al primado por la proteína de la célula anfitriona con actividad furina TMPRSS2, empieza rápidamente la detección del ssRNA(+) del coronavirus. Para ello la célula dispone de los receptores citosólicos RIG-I-like receptors (RLRs) RIG-I y MDA5 y los Toll-like receptors de las membranas endosomales (TLR3, TLR7, y TLR8) que perciben además los intermediarios de replicación en forma de dsRNA virales. La interacción de estos receptores de patrones moleculares virales gracias a los intermediarios de transducción MAVS, MyD88, y TRIF inicia dos cascadas de señalización independientes que llevan al aparición de dos factores de transcripción. Para ello es necesario que se activen varias kinasas de IκB (IKKs), llamadas kinasa 1 de unión a TANK (TBK1)/inhibidor de la kinasa κB épsilon (IKKε) e IKKα/β/ɣ (IKKɣ se conoce también como NEMO). Por un lado IKKα/β/ɣ fosforilan al inhibidor de NF-κB (IκB), liberándose entonces de NF-κB y activando la expresión de citocinas pro-inflamatorias. Por otro lado, TBK1 fosforila a IRF3 convirtiéndolo entonces en el factor de transcripción que se transloca al núcleo e inicia la expresión de genes asociados al los interferones de tipos I y III.
En paralelo, TLR3 es activado por dsRNA que induce la señalización de IRF3 y NF-κB a través de cascadas de señalización de interferón‐β (TRIF)‐TBK1/IKKε que contiene el dominio TIR y que induce el adaptador, lo que da como resultado la producción de IFN de tipo I y III y otras citocinas pro-inflamatorias. Aún no se ha dilucidado la implicación de la vía de detección del ADN citosólico del estimulador cGAS de la señalización de genes de IFN (STING) en el reconocimiento de coronavirus. STING es activado por el segundo mensajero 2ʹ−3ʹcGAMP producido por cGAS al ser activado por ADN. Posteriormente, STING recluta TBK1 y con ello sigue toda la vía que hemos visto anteriormente,
Luego, las vías de señalización RIG-I/MDA-5-MAVS, TLR3-TRIF y cGAS-STING convergen en TBK1/IKKε, que cataliza la fosforilación de IRF3 y la posterior transcripción de los IFN de tipo I y III. Los IFN secretados de tipo I y III se unen a sus receptores y luego activan los transductores de señal kinasa de Janus (JK) y los activadores de la señalización de transcripción (STAT) para impulsar la expresión de genes estimulados por IFN (ISG), que pueden iniciar estados antivirales al suprimir la replicación y propagación viral, activar las células inmunitarias y provocar la muerte de las células infectadas.
TOM70 y el transporte de proteínas a la mitocondria
La célula eucariota emergió después de una integración entre una arquea y su simbionte bacteriano. La célula huésped y el simbionte, que se convirtió en mitocondria, han co-evolucionado durante tanto tiempo que son completamente dependientes el uno del otro. Por un lado, las células modernas dependen de las mitocondrias para la producción eficiente de ATP y cofactores vitales, como los grupos de hierro-azufre y el hemo. Por otro lado, las mitocondrias han renunciado a su autonomía para la producción de lípidos y proteínas y ello recae en sus células hospedadoras. Ello crea una interdependencia constante entre las mitocondrias, el citosol y todos los demás orgánulos a través del flujo de metabolitos, la participación en vías de señalización comunes y el transporte de proteínas recién sintetizadas destinadas a las mitocondrias, en forma de proteínas precursoras. En el desarrollo de la relación endosimbiótica, la bacteria retuvo su cromosoma en forma de anillo pero perdió la mayoría de sus genes. En todos los eucariotas, aproximadamente el 99% de las proteínas mitocondriales están codificadas por el ADN nuclear y sintetizadas por los ribosomas citosólicos. El proteoma mitocondrial humano comprende aproximadamente 1500 proteínas diferentes.
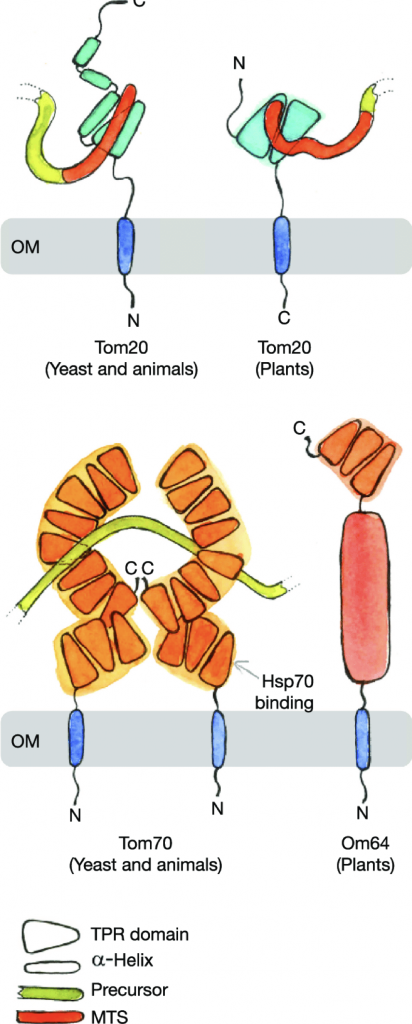
Importar esas proteínas en varias ubicaciones mitocondriales es una de las tareas más desafiantes de la biología celular. El primer paso de la focalización implica el reconocimiento y movimiento direccional de la proteína (o su ARNm codificante) desde el citosol a la superficie del orgánulo correcto. A continuación, la proteína se puede entregar a la maquinaria responsable de su integración en las membranas o su paso a los compartimentos acuosos del orgánulo mediante translocación a través de canales o poros de proteínas. Todo el proceso de focalización y traslado a las mitocondrias constituye la importación de proteínas mitocondriales. Las proteínas que deben importarse a diferentes compartimentos contienen señales de orientación específicas que son reconocidas tanto por las maquinarias de orientación como por las de translocación.
La translocación de proteínas a las mitocondrias está bien estudiada y está claro que existen muchas vías diferentes para ayudar a la correcta translocación e integración de proteínas a sus subcompartimentos mitocondriales. Todos ellos comienzan en la translocasa del complejo de la membrana externa (TOM). El complejo TOM consta de la proteína de barril β Tom40 que forma un poro de translocación, las proteínas receptoras Tom20 y Tom70 que reconocen señales de direccionamiento en los proteínas precursoras, Tom22 que puede reconocer estos precursores y también desempeña un papel estructural, y las pequeñas subunidades Tom5, 6, y 7 que median el complejo ensamblaje.
Casi todos los complejos de importación de proteínas a los orgánulos membranosos contienen una repetición tetratricopeptídica (TPR) para mejorar la eficacia de la translocación de proteínas. Por tanto, vale la pena para echar un vistazo más de cerca a las funciones y características estructurales de los dominios TPR que se encuentran en Tom70 y sus análogos funcionales en otras membranas. Un dominio TPR consta de hasta 20 repeticiones tetratricopeptídicas, cada una de las cuales comprende 34 aminoácidos que adoptan un motivo hélice-giro-hélice y u ángulo de empaquetadura de aproximadamente 24º. Por lo tanto, los motivos TPR apilados inducen una superhélice que forma un canal o poro y que también es la estructura predominante de Tom70.
Además de esa función en la importación de proteínas, TOM70 es un elemento crítico en una cascada de señales que conduce a una respuesta inmune innata específicamente al ARN viral. De hecho cuando RIG-I sufre un cambio conformacional al reconocer el ARN viral y se asocia con MAVS, esto provoca que MAVS se asocie con TOM70. En realidad es esta participación de TOM70 lo que inicia el reclutamiento de Hsp90, TBK1 e IRF3. Si vamos a la estructura íntima del proceso, dentro de este complejo, el motivo EEVD C-terminal de Hsp90 se une al dominio de tipo abrazadera TPR N-terminal de TOM70. Posteriormente, el IRF3 es fosforilado por TBK1, lo que conduce a su disociación del complejo y así IRF3 fosforilado se transloca al núcleo y promueve la transcripción de genes. Además, la unión de MAVS a TOM70 también puede desencadenar la señalización de NF-κB y la apoptosis dependiente de Bax.
¿Cuáles son las estrategias que hacen que el RNA del SARS-CoV-2 perdure en la célula y no inicie la respuesta innata?
En primer lugar la proteína no estructural nsp16 metila la cabeza 5’ del RNA viral haciéndole que se asemeje al RNA humano y así facilitándole el escape del sensor MDA5. La nsp3 produce una proteasa como la papaína (PLpro) que junto con las ORF3b y ORF6 interfieren con la fosforilación y por consiguiente translocación nuclear de IRF3. Además ORF6 limita el efecto de la respuesta a interferones bloqueando en transporte nuclear del la STAT1 e impidiendo la expresión de ISG. Por otro lado, el dominio de proteasa similar a la papaína de los virus SARS puede actuar como antagonista de los IFN dirigiéndose a STING, lo que sugiere que la vía cGAS-STING debería desempeñar un papel vital en la defensa contra ciertos coronavirus.
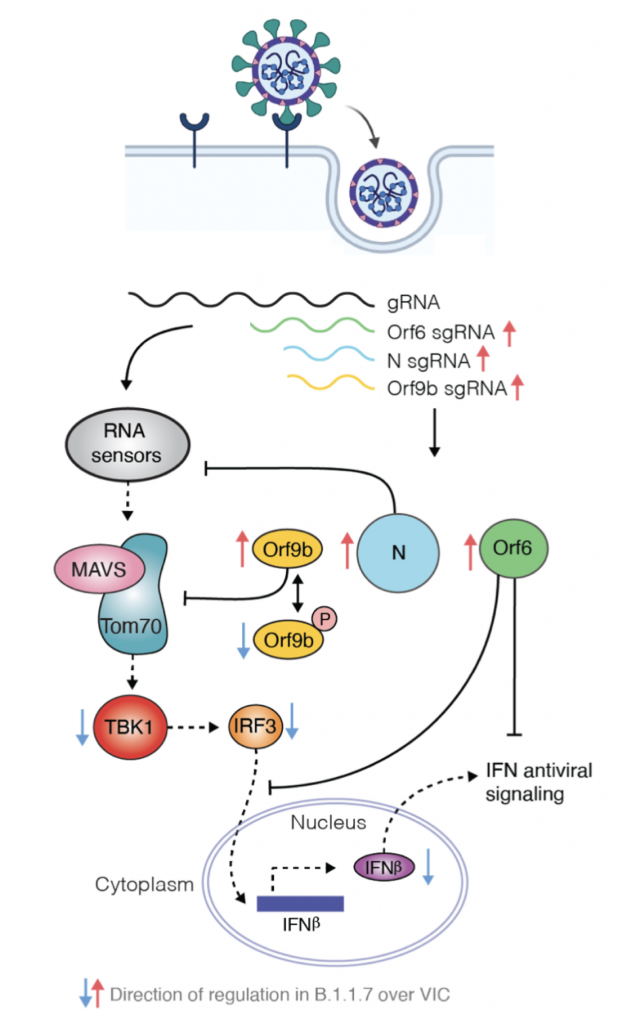
Una de las mutaciones que se han descrito ya en la variante alpha (la británica, B.1.1.7) afecta a una proteína accesoria, en concreto ORF9b. Curiosamente estas mutaciones no se traducen en cambios estructurales o de función de la proteína sino lo que consiguen es que la proteína se exprese hasta 18 veces más en la célula. La proteína accesoria ORF9b se acumula rápidamente tras la infección y también bloquea la producción de IFN-β al interferir con la actividad de las proteína mitocondrial MAVS, que es un adaptador esencial para el reconocimiento por RIG-I/MDA5 y su señalización antiviral. Además, ORF9 conduce a la degradación de MAVS impidiendo la señalización inducida por TBK1-IRF3 y específicamente sobre NEMO para evitar la activación de NF-κB. La secuencia de actuación podría ser la siguiente: ORF9b se expresa ectópicamente en la mitocondria donde interactúa con TOM70 y MAVS provocando cambios mORFológicos y funcionales en estas y la translocación de proteínas en las membranas de la mitocondria y de estas al citosol. Al proteína TRAF añadiría colas de ubiquitina a NEMO lo que facilitaría la interacción entre NEMO y MAVS y el inicio de la señalización. En ese mismo momento es cuando ORF9b ya podría actuar sobre NEMO inhibiéndolo. A diferencia de los otros tres mecanismos, ORF9b no se comparte con otros coronavirus (excepto por SARS-CoV-1, con quien comparte un 74% de homología) por lo que es un rasgo singular de SARS-CoV-2 y único en su manera de evitar la repuesta inmunitaria antiviral. Por tanto, la sobreexpresión de SARS‐CoV‐2 ORF9b inhibe la producción de IFN de tipo I y III inducida tras la activación de RIG-I/MDA5–MAVS, TLR3–TRIF y cGAS–STING.
Genética, estructura, función y evolución de la proteína accesoria ORF9b
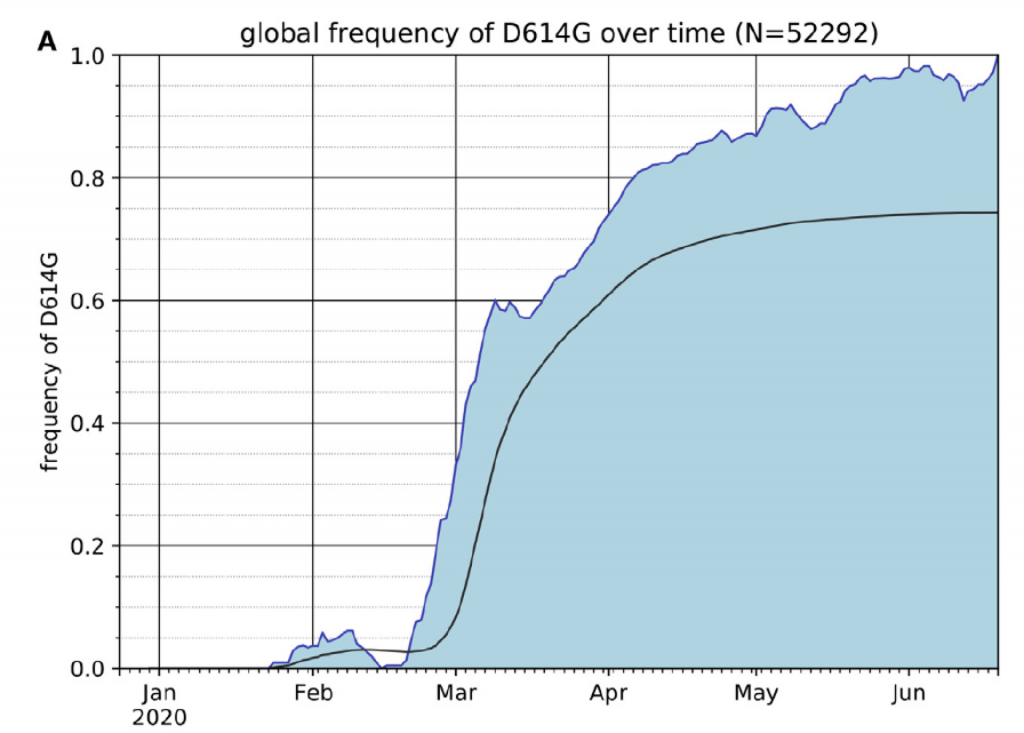
La estructura del genoma del SARS-CoV-2 indica que hay 14 ORF (open reading frame, cualquier parte del genoma que puede ser traducido en una proteína al tener codones de inicio y de stop). Algunas de estas ORFs se solapan en el genoma del virus optimizando así es reducido material genético y consiguiendo que se codifiquen para más proteínas de las que daría su limitado número de pares de bases. Las ORF1a y ORF1b son las más largas y se traducen en dos poliproteínas PP1A y PP1AB que contienen las proteasas PLpro y 3CLpro que convierten esas dos poliproteínas en 16 proteínas no estructurales que entre otras funciones tienen actividad replicasa y transcriptasa y son esenciales para la replicación y maduración viral. Las otras ORFs 2, 4, 5, y 9a codifican para las 4 proteínas estructurales principales de los coronavirus, a saber, Spike (S), Envelope (E), Membrane (M) y Nucleocapsid (N), respectivamente. Y ahora vienen las diferencias fundamentales entre los distintos coronavirus, pues las otras ORFs codifican para proteínas accesorias sin papel en la replicación viral como son ORF3a, ORF3b, ORF6, ORF7a, ORF7b, ORF8a y ORF8b y por consiguiente están presentes o no en diversos coronavirus. Por ejemplo la ORF3b no se expresa en el SARS-CoV-2 y la ORF9b no estaba presente en la cepa original surgida en Wuhan, sino que aparece por primera vez en la variante D614G, que se hizo dominante en el mundo en la primavera del año 2020.
Para ser más precisos la ORF9b se sitúa dentro de la ORF9a y que codifica para la proteína estructural N (por eso se clasifica a la ORF9b como una ORF alternativa). Esta nueva proteína ORF9b se expresa ampliamente en las células infectadas, tiene 98 aminoácidos y la cristalografía muestra que el homodímero presenta unas características estructurales con nuevos dominios de plegamientos de tipo beta con una superficie anfipática y un núcleo hidrofóbico que interacciona muy bien con lípidos. Habida cuenta que ORF9b actúa sobre el signalosoma mitocondrial MAVS/TRAF3/TRAF6 el análisis comparativo de interacciones virus-proteínas del anfitrión mostró que la diana de interacción de ORF9b es la proteína de la membrana externa mitocondrial TOM70.
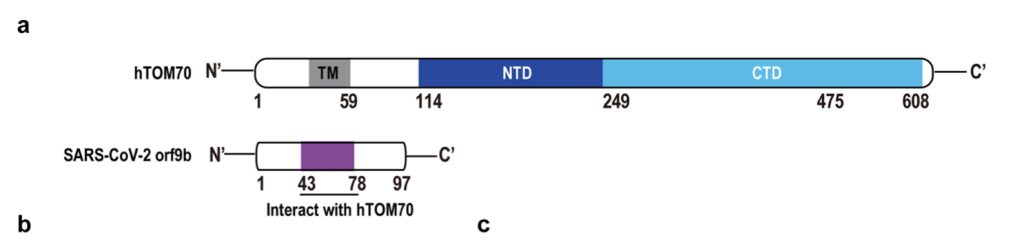
Como ya hemos dicho, esta proteína TOM70 forma parte de un complejo transmembranario TOM con actividad translocasa y por consiguiente implicado en la tranlocación de proteínas desde el retículo endoplasmático rugoso al interior mitocondrial y además TOM70 juega un papel muy importante en la activación de la respuesta antiviral pues es un adaptador esencial para la señalización del MAVS a TBK1/IRF3. Estructuralmente TOM70 consta de n repeticiones de dominios de tipo TPRs (tetratricopeptide repeats). El TPR de la región N-terminal de TOM70 se une a la C-terminal de Hsp90, resultando en el reclutamiento y formación del complejo Hsp90/TBK1/IRF3 en la mitocondria. Así, IRF3 es fosforilado y translocado al núcleo para actuar como factor de transcripción. A nivel de microscopía crio-electrónica de ~3.1 Å de resolución se ha demostrado que ORF9b ocupa un dominio hidrofóbico en la región TPR C-terminal, que es la zona por donde TOM70 se une a las proteínas para importarlas. Además, la cristalografía del homodímero de ORF9b indica que una porción central de ORF9b ocupa el bolsillo profundo en el dominio C-terminal de TOM70 (CTD) y adopta una conformación helicoidal sorprendentemente diferente de la estructura rica en hojas β del homodímero ORF9b. Las interacciones entre ORF9b y TOM70 CTD son principalmente hidrófobas y distintas de la interacción electrostática entre el motivo EEVD de la proteína de choque térmico 90 (Hsp90) y el dominio N-terminal de TOM70 (NTD). Luego ya tenemos evidencia experimental que respalda que la proteína ORF9b del SARS-CoV-2 inhibe alostéricamente la unión entre TOM70 y Hsp90, lo que en consecuencia puede socavar el reclutamiento de Hsp90 a TBK1/IRF3 y, por lo tanto, en última instancia, debilita la cascada de señalización para expresión de interferones de tipo 1.
Repercusiones de las nuevas variantes
La infección por la variante alpha se ha asociado con mayores tasas de contagio y mayor gravedad clínica. La variante alpha se define por una constelación de 23 mutaciones: 17 que alteran la secuencia de proteínas (14 no mutaciones sinónimas y 3 deleciones) y 6 mutaciones sinónimas. Los cambios se concentran en Spike, que facilita la entrada viral a través de la interacción con el ser receptor ACE humano. Esto ha llevado al campo a enfocarse en comprender estas variantes, pero como hemos dicho la principio del ensayo, hay otras. De hecho las mutaciones que definen la variante de preocupación (VOC) fuera de Spike sugieren que la adaptación independiente de Spike al anfitrión puede contribuir a la ventaja de transmisión de alpha. La mayoría de los cambios de codificación de alpha se asignan a proteínas no estructurales nsp3, nsp6, la proteína accesoria ORF8 y N, habiendo demostrado todas su capacidad de modular la respuesta inmune innata.
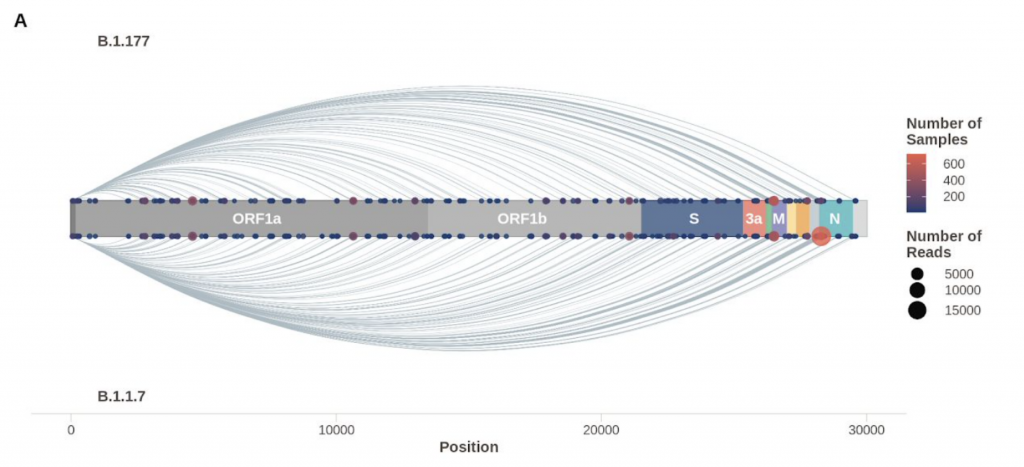
En consonancia con la resistencia al IFN, frente a los aislados de la primera ola, la variante alpha es menos sensible al tratamiento previo con IFN-β en un amplio rango de dosis, lo que sugiere que la infección por alpha no solo induce menos IFN-β sino que que también es menos sensible a sus efectos. La mutación D614G se ha asociado con resistencia a una variedad de interferones de tipos I y III en varios linajes de SARS-CoV-2, y contribuye a mejorar la evasión de IFN de alpha. Ahora tenemos datos que revelan cómo el linaje SARS-CoV-2 alpha se ha adaptado al huésped mejorando el antagonismo de la respuesta inmune innata. Sorprendentemente se ha caracterizado una nueva forma de adaptación que viene más que de la mutación estructural en aumentar específicamente la síntesis de ARN subgenómico y la expresión de antagonistas víricos innatos clave como ORF9b y ORF6 y N. Esta observación notable y novedosa sugiere la evolución de las secuencias de nucleótidos de alpha que modulan la producción de sgRNA específica y la selección para una mayor síntesis de sgRNA y expresión de proteínas, en lugar de la selección de cambios en la codificación de proteínas para alterar o mejorar la función del antagonista viral.
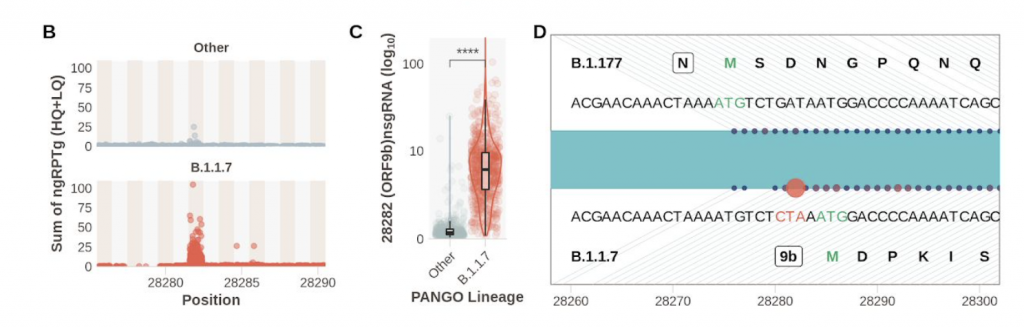
Así, los niveles de expresión de proteínas pueden tener un impacto significativo en la interacción virus-huésped e importantes implicaciones para el manejo de la pandemia en curso. Se espera que la expansión de los esfuerzos de secuenciación en curso para monitorear los niveles de ARN subgenómico sea fundamental en la identificación de futuras variantes de interés del SARS-CoV-2. Estos hallazgos resaltan la importancia de estudiar los cambios fuera de Spike para comprender el fenotipo de la variante alpha, otras variantes actuales y variantes futuras, y enfatizar la importancia de la evasión inmune innata en el proceso continuo de adaptación del SARS-CoV-2 a un nuevo anfitrión. Podemos asumir que esta mejora es una consecuencia directa de una mutación de triple nucleótido en la nucleocápside (28280: GAT> CAT, D3L) que crea una secuencia de tipo reguladora de la transcripción complementaria a una región 3 ‘del líder genómico. Estos hallazgos proporcionan una visión única de la biología de la variante alpha y respaldan el seguimiento de los perfiles de sgRNA en los datos de secuencia para evaluar las posibles variantes emergentes de interés.
Conclusión
SARS-CoV-2 usa diversas estrategias para neutralizar la respuesta innata antiviral y el inicio de la respuesta inmunitaria innata más allá de modificar la proteína Spike. La proteína codificada por ORF9b antagoniza a la interacción del translocador mitocondrial TOM70 con otros partners como hsp90 lo que al final provoca que la participación mitocondrial en la respuesta de interferones quede bloqueada. Hablamos de mutaciones que no llevan a cambios estructurales de ORF9b sino en regular al alza los niveles de expresión de la proteína. Por ello el análisis de expresión de sgRNA debe realizarse en todas las plataformas de vigilancia genómica compatibles para dar una lectura instantánea de los perfiles de expresión alterados en las variantes emergentes de SARS-CoV-2. Esto utilizaría los datos existentes para complementar los métodos epidemiológicos y filodinámicos, y proporcionaría una alerta temprana de las variantes que podrían ser motivo de preocupación con respecto a una mayor transmisibilidad y / o gravedad de la enfermedad.
Referencias
- Thorne, L. G., et al. (2021). Evolution of enhanced innate immune evasion by the SARS-CoV-2 B.1.1.7 UK variant. BioRxiv, 2021.06.06.446826. https://doi.org/10.1101/2021.06.06.446826
- Alexander, J., et al. (2021). Altered Subgenomic RNA Expression in SARS-CoV-2 B.1.1.7 Infections. BioRxiv, 2021.03.02.433156. https://doi.org/10.1101/2021.03.02.433156
- Huang, Y., et al. (2020). Structural and functional properties of SARS-CoV-2 spike protein: potential antivirus drug development for COVID-19. Acta Pharmacologica Sinica, 41(9), 1141–1149. https://doi.org/10.1038/s41401-020-0485-4
- Mallapaty, S. (2021). How a rampant coronavirus variant blunts our immune defences. Nature, 2021. https://doi.org/10.1038/d41586-021-01540-8
- West, A. P., et al. (2011). Mitochondria in innate immune responses. Nature Reviews Immunology, 11(6), 389–402. https://doi.org/10.1038/nri2975
- Melino, G., et al. (2008). Itch: A HECT-type E3 ligase regulating immunity, skin and cancer. Cell Death and Differentiation, 15(7), 1103–1112. https://doi.org/10.1038/cdd.2008.60
- Gordon, D. E., et al. (2020). A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature, 583(7816), 459–468. https://doi.org/10.1038/s41586-020-2286-9
- Gao, X., Zhu, K., et al. (2021). Crystal structure of SARS-CoV-2 Orf9b in complex with human TOM70 suggests unusual virus-host interactions. Nature Communications, 12(1), 1–9. https://doi.org/10.1038/s41467-021-23118-8
- Han, L., et al. (2021). SARS-CoV-2 ORF9b antagonizes type I and III interferons by targeting multiple components of the RIG-I/MDA-5–MAVS, TLR3–TRIF, and cGAS–STING signaling pathways. Journal of Medical Virology, April, 1–14. https://doi.org/10.1002/jmv.27050
- Wu, J., et al. (2021). SARS-CoV-2 ORF9b inhibits RIG-I-MAVS antiviral signaling by interrupting K63-linked ubiquitination of NEMO. Cell Reports, 34(7), 108761. https://doi.org/10.1016/j.celrep.2021.108761
- Kreimendahl, S., & Rassow, J. (2020). The mitochondrial outer membrane protein tom70—mediator in protein traffic, membrane contact sites and innate immunity. International Journal of Molecular Sciences, 21(19), 1–32. https://doi.org/10.3390/ijms21197262
- Bykov, Y. S., et al. (2020). Cytosolic Events in the Biogenesis of Mitochondrial Proteins. Trends in Biochemical Sciences, 45(8), 650–667. https://doi.org/10.1016/j.tibs.2020.04.001
